| 多国及时发现变异新冠病毒,基因测序功不可没! | ||
| 谁也没有预料到,2020年是如此不寻常的一年。突如其来的新冠疫情,打破了以往的生活。 好在我们有,医护人员逆风而上救死扶伤,科研人员争分夺秒解析病毒、研发疫苗,各行各业乃至个人众志成城、共同战“疫”,我们相信,一切恢复正轨指日可待! 但是现在,我们仍然不能放松警惕! |
||
| 传染性更强的变异新冠病毒出现!世卫组织建议各国增加对新冠病毒的常规测序! | ||
| 据央视新闻报道,当地时间12月22日,世卫组织发布英国上报的有关变异新冠病毒的相关信息。12月14日,英国向世卫组织报告,通过病毒基因测序发现一种新的新冠病毒变体。初步分析表明,该变体更易在人与人之间传播,估计传染性增加40-70%,传播指数增加0.4在1.5-1.7之间[1]。 | ||
| 世卫组织建议各国在可能的情况下增加对新冠病毒的常规测序,并分享病毒基因序列数据,尤其要报告是否发现了相同的变异病毒。所有国家都要评估当地病毒传播水平,并采取适当的预防和控制措施[1]。 | ||
| 从最初的新冠肺炎确诊标准之一,到目前成为疫情防控的必要手段,高通量测序技术(NGS)在新冠病毒检测中可以发挥哪些作用呢? | ||
| 带着这个问题,我们一起来深度了解一下,高通量测序在新冠病毒检测中的应用。 |
||
| 依靠NGS技术获得病毒的基因组序列,揭开新冠病毒的真面目! | ||
| 通过NGS技术可以直接获得病毒的全基因组序列,进而全面分析病毒基因,为突发疫情的防控、临床治疗和后续研究提供帮助。 | ||
| 2020年2月3日,复旦大学张永振教授,在Nature发表题为A new coronavirus associated with human respiratory disease in China的文章,较早地向世界公布了新冠病毒的RNA序列,这一举措也为应对COVID-19奠定了重要的基础。该研究取一名41岁的男性患者的200 μL肺泡灌洗液(BALF)后提取总RNA,使用Illumina MiniSeq(PE 150)进行深度宏转录组测序,确定了该病毒的基因组。同时进行系统进化分析,发现与一组来自于中国蝙蝠SARS样冠状病毒基因组相似度达到89.1%。作者使用了SMARTer Stranded Total RNA-Seq Kit v2- Pico Input Mammalian制备宏转录组文库,通过核心的ZapR v2、Rprobes v2技术在建库过程中去除了人源rRNA! |
||
| 通过对新冠病毒进化研究,了解新冠病毒的“作案特征”和“行动路线” | ||
| 通过构建进化树揭示其病原相关特性、分析病毒的进化来源等,对于设计有效的疾病控制和预防策略相当重要。在《新型冠状病毒肺炎防控方案(第七版)》文件中指出,各地发生的首例和感染来源不明的病例以及环境监测发现的阳性标本,应当开展基因测序等溯源工作。 | ||
| 2020年4月16日,广东省疾病预防控制中心柯昌文教授和牛津大学Oliver G Pybus共同通讯在Cell发表题为“Genomic epidemiology of SARS-CoV-2 in Guangdong Province, China”的文章,该研究结合了宏基因组测序和平铺扩增子方法,从广东省感染个体中产生了53个基因组,进行系统发育分析,结合流行病学信息,分析新冠病毒的进化来源等。该研究结果可能为在其他地区实施和解释COVID-19基因组检测提供有价值的信息。其中,同样使用了SMARTer Stranded Total RNA-Seq Kit v2- Pico Input Mammalian对鼻拭子、咽拭子和痰液等来源的样本,制备宏转录组文库! | ||
| 这里允许小编地向大家介绍一下,频获众多科研人员青睐的文库构建试剂盒 |
||
| ↓ |
||
| SMARTer Stranded Total RNA-Seq Kit v2 - Pico Input Mammalian(Code: 634411) | ||
| 我们知道,在使用鼻拭子、咽拭子、肺泡灌洗液等临床样本进行病原微生物NGS分析时,往往会遇到样本含量少、样本背景复杂、宿主核酸干扰大、建库过程PCR扩增不均一等问题,给正确分析病毒基因组信息造成困难。 | ||
| 而SMARTer Stranded Total RNA-Seq Kit v2 - Pico Input Mammalian,这么受大家喜欢,就是因为它能很好的应对这些问题: |
||
| 它专为低质量样本设计,无需进行rRNA前处理操作,可直接以250 pg-10 ng 的total RNA起始,6小时内即可制备高质量的Illumina文库,已被大量用于新冠病原微生物的检测分析! | ||
| 参考文献: | ||
| 1. Chen, Long, and Li Zhong. “Genomics functional analysis and drug screening ofSARS-CoV-2.” Genes & diseases, 10.1016 (2020). 2. Thair, Simone et al. “Transcriptomic Similarities and Differences in Host Response between SARS-CoV-2 and Other Viral Infections.” medRxiv (2020). 3. Pfefferle, Susanne et al. “Complete Genome Sequence of a SARS-CoV-2 Strain Isolated in Northern Germany.” Microbiology resource announcements vol. 9,23 (2020). |
||
| 面对不断变异的病毒,NGS技术在疫情防控工作中发挥重要作用! | ||
| 病毒变异是病毒复制过程中发生的常见现象。新冠病毒作为RNA病毒,变异相对会更加频繁。 | ||
| NGS可以动态跟踪病毒变异,不仅可以进行针对性预案,也可以为后期有效的治疗提供基础性研究依据。 | ||
| 但是,对于需要如此高正确度检测时,一旦出现测序错误、或者PCR偏差,便会产生大量假阳性结果,很可能就影响结果的正确性。面对这样的情况,当然不能放任不管,刚好这里有一个锦囊妙计,供您参考:通过添加特别的分子签UMI来有效改善这一问题。 | ||
|
||
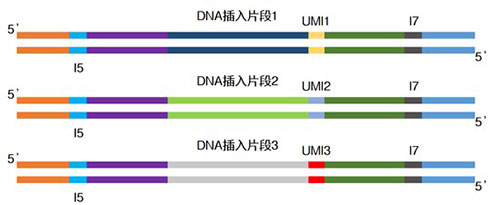 |
||
| 举个例子 | ||
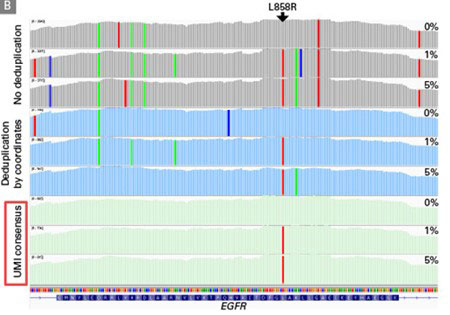 |
||
| 如图所示,使用未去重或者仅用坐标法去重的文件分析,在预期的EGFR L858R突变附近观察到大量的低频及随机等位基因频率突变。而用UMI校正、过滤后的一致性序列reads,清除了假阳性突变,得到了预期的突变分析结果! | ||
| 有了妙计,还需要能上手的真家伙。这不,它来了… | ||
| Takara特别优化推出了接头上添加分子标签的全新产品:SMARTer Stranded Total RNA-Seq Kit v3 - Pico Input Mammalian(Code No. 634485)。 | ||
| 火眼金睛,发现“真”病毒变异 | ||
| 在文库制备过程中,通过反转录步骤(在PCR扩增之前)添加UMI,可以灵敏地识别PCR重复片段并校正PCR扩增错误,从而进行正确的样本数据回溯,提供更可靠的RNA-Seq分析! | ||
| 致敬“前浪”,同样无惧困难样本 | ||
| 该产品保留了前一代产品能分析质量较差样本和无需核糖体RNA前处理的特点,支持皮克级总RNA起始,快速高效的构建测序文库! | ||
| 相信SMARTer Stranded Total RNA-Seq Kit v3 - Pico Input Mammalian依仗自身过硬的特点,可以为新冠病毒变异监测贡献一份力量! | ||
| 参考信息来源: | ||
| 1. 央视新闻,2020年12月22日 | ||
| 最后温馨提醒大家:进入冬季以后,新冠病毒疫情不断反复,又正值新年之际,因此注意佩戴好口罩,养成良好的卫生习惯,愿大家都身体健康。 | ||
页面更新:2020-12-31 07:59:22