| 使用10 kb或更大的长链通过多重PCR进行长读长NGS分析 | ||
| 多重PCR是一种通过在单个PCR反应体系中使用多对引物,同时扩增多个基因区域的方法。多重PCR可以节省试剂和耗材从而降低成本,并且可以通过同时检测提高速度,能够更有效地利用珍贵的样品。 然而,多重PCR容易产生非特异性扩增,存在引物设计复杂、反应优化等缺点。 在这项研究中,我们使用具有高特异性的PCR酶PrimeSTAR® LongSeq DNA Polymerase(Code No. R055S/A)来评估长链多重PCR的可行性(实验例1)。 R055也是一种高保真DNA聚合酶,适用于对保真性要求较高的下一代测序(NGS)分析。 因此,我们对使用长读长测序(Oxford Nanopore Technologies)分析了通过长链多重PCR获得的扩增产物(实验例2)。 |
||
 |
||
| 图1. 实验流程 | ||
| ■ 实验例1:20重长链多重PCR实验 | ||
| ● 方法 ·酶的使用: PrimeSTAR® LongSeq DNA Polymerase 适用于长链扩增的其他公司PCR酶 ·模板: 人类基因组 DNA ·片段: 所有20个片段(长度:10-12 kb,GC 含量:35-60%) ·反应体系: 在50μl反应体系中添加100ng的人类基因组DNA作为模板,各引物的最终浓度为0.2μM。 ·PCR条件: PrimeSTAR® LongSeq DNA Polymerase |
||
| 94℃ 1 min | ||
| ↓ |
||
| 98℃ 10 sec. | 30 cycles | |
| 68℃ 30 sec/kb | ||
| *其他公司的PCR酶:说明书推荐的PCR条件 *PCR反应在N=2条件下进行 仪器:Clontech PCR Thermal Cycler GP(Code No. WN400) |
||
| ● 结果 | ||
 |
||
| 图2. 20重片段的多重PCR 在纯化多重PCR产物后,使用4200 TapeStation System(Agilent Technologies)进行分析。结果表明使用R055可以特异性地扩增10~12 kb附近的片段。 |
||
| (Takara Bio Inc.比较结果) | ||
| ■ 实验例2:NGS 分析 | ||
| ● 方法 在实验例1中确认有三个酶可以扩增10~12 kb附近的片段,使用Native Barcoding Kit 96 V14(Oxford Nanopore Technologies)构建NGS文库,并使用GridION(Oxford Nanopore Technologies)和R10.4.1 flow cells (Oxford Nanopore Technologies)进行NGS分析。对长读长NGS获得的碱基序列进行了读取数一致的分析。此外,在本实验中未优化引物浓度,而是使用了等摩尔混合的引物组。 |
||
 |
||
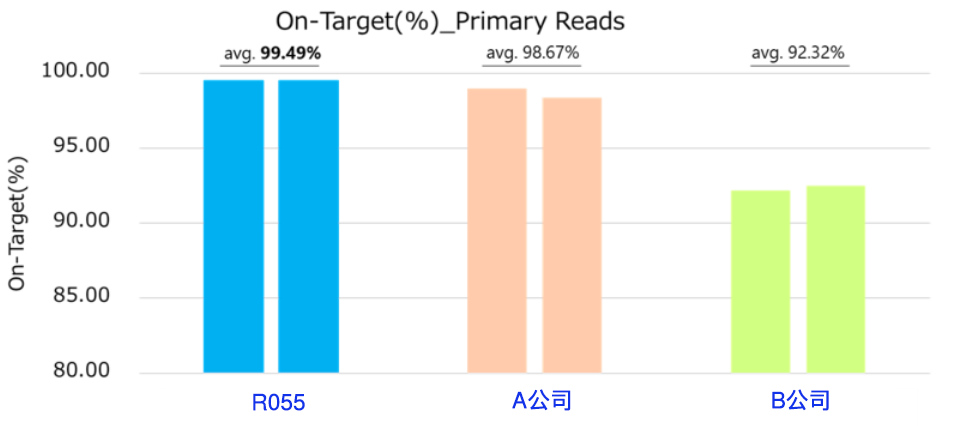
| 图3. 各酶的靶向区域覆盖(On-Target) | ||
| 根据生成的序列读数,计算了各酶的靶向区域覆盖。 使用R055扩增的产物显示出99%以上的靶标覆盖率,表明R055在长链多重PCR中具有较高的特异性扩增。 | ||
 |
||
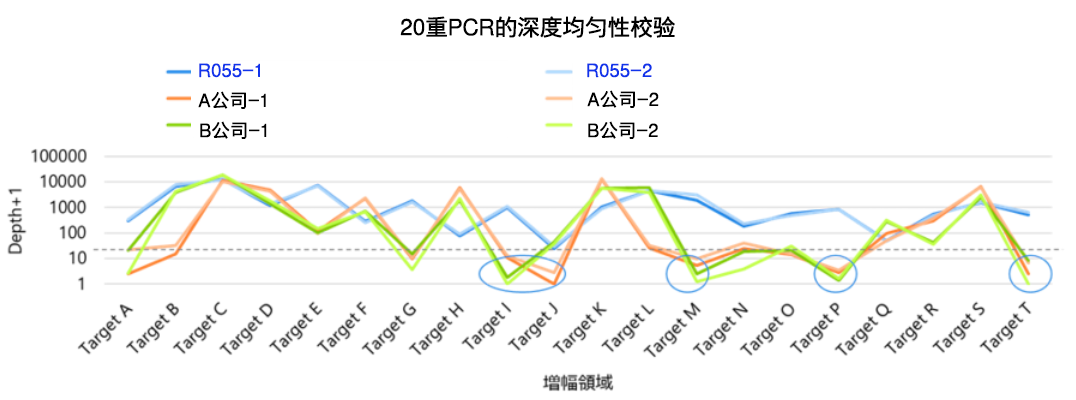
| 图4. 20重PCR的深度均匀性校验 | ||
| 在多重PCR中,各PCR靶标的扩增效率差异是一个问题。 我们确认了在各个PCR 酶的扩增产物中是否含有20个靶标的序列。 通过使用R055可以用于实现更均匀的扩增。相比之下,其他公司的PCR酶在多个靶标上没有产生深度或产生极低的深度,但使用R055可以获得更少偏差的均匀深度。对于难以扩增的区域,即使增加测序量也难以改善,测序成本会上升。 使用R055可以减少多重反应PCR的优化工作,并能够以更小的测序量进行分析。 | ||
| (Takara Bio Inc.比较结果) | ||
 |
||
 |
||
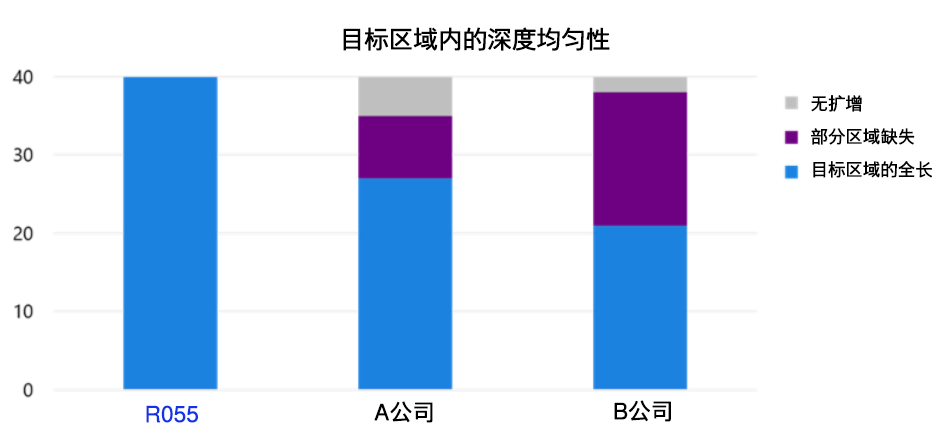
| 图4. 目标区域全长的扩增确认 | ||
| 使用Integrative Genomics Viewer(IGV)来确认各酶的多重PCR扩增产物中是否包含20个靶标序列。R055可以在所有20个靶标上进行全长扩增,但其他公司的PCR酶缺少部分区域,只能在68-78%的靶标上进行全长扩增。 *未获得靶标L读数的区域属于用于验证的人基因组DNA中已知的大缺失区域(976 bp)。 |
||
| (Takara Bio Inc.比较结果) | ||
| ● 结论 在本次验证中,评估了六种PCR酶,并确认了三种PCR酶可以通过20个靶标的多重PCR进行特异性扩增。在三种PCR酶中,R055是唯一获得所有20个靶标区域全长序列的PCR酶。 研究表明,PCR酶的选择对于通过长链多重PCR对靶标区域进行全长NGS分析至关重要。 |
||

 京公网安备 110114000405号
京公网安备 110114000405号