| 1+1>2,Trekker探秘“单细胞+空间转录组技术” |
| |
细胞作为生命活动的基本单元,其基因表达调控网络与细胞间互作机制是生命科学的重要研究课题。单细胞测序技术虽能在单细胞水平解析基因表达状态,却因破坏细胞在组织中的空间定位,难以揭示组织微环境中细胞互作及功能协同的真相。
空间转录组技术的出现,填补了这一关键空白。通过整合转录组测序与空间定位,实现了组织中细胞类型、基因表达及空间分布的同步解析,为研究细胞在微环境中的功能提供了关键支撑。但常规空间转录组的分辨率局限往往导致无法精确定位至单个细胞,犹如雾里观花。
因此现在很多技术不得不花费精力用复杂的算法整合单个细胞和空间数据,并试图调和不同技术固有的测量偏差。这恰恰凸显了一个更直接的需求:为何不直接测量单个细胞在原生环境中的空间位置? |
| |
|
2024年,Nature刊载文章《Slide-tags enables single-nucleus barcoding for multimodal spatial genomics》。研发者开发Slide-tags技术,其中完整组织切片内的单个细胞核用空间条形码寡核苷酸标记,这些寡核苷酸来源于具有已知位置的DNA条形码微珠,这些被标记的细胞核可以进行多种单细胞核组学分析,适用于大部分单细胞测量技术,为空间组学的研究提供了一个通用的平台。 |
| 基于这项技术转化而来的Trekker Single-Cell Spatial Mapping Kit,实现了真正意义上的单细胞空间转录组分析——通过“空间定位 + 基因解码”的协同设计,直接对单细胞数据进行位置解析。 |
| |
|
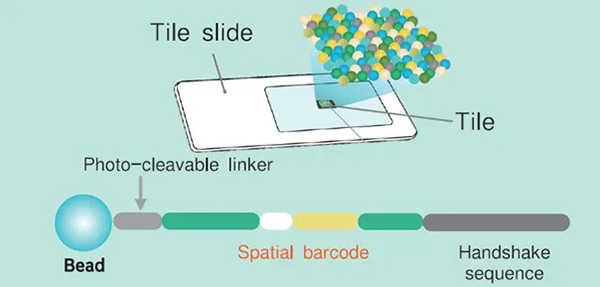
| ■ 微珠布阵 |
| 将带有DNA条形码的10 μm单层微珠(Beads)有序平铺于玻片(Tile),形成空间坐标已知的“捕获阵列”。 |
 |
| |
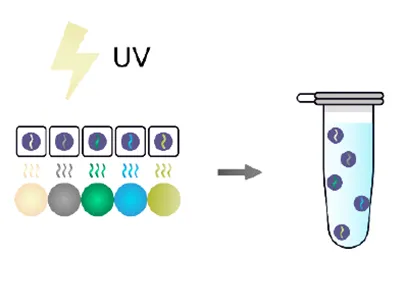
| ■ UV照射 |
| 在玻片上放置组织冷冻切片,通过紫外照射,微珠上的DNA条形码被触发释放,吸附着于细胞核上,相当于为每个细胞核赋予了独一无二的“空间坐标码”。该序列可以通过单细胞分析系统进行测序,其中微珠条状条形码可以与mRNA等待测分子同时进行序列分析。 |
 |
| |
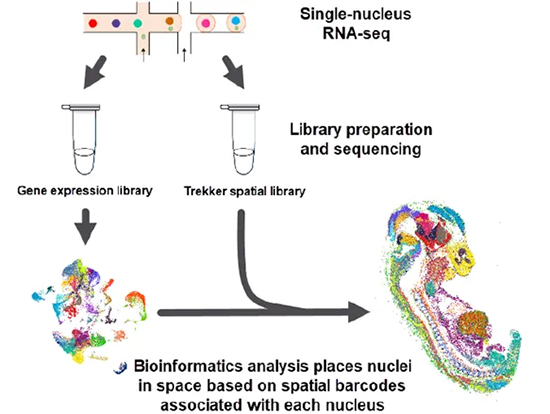
| ■ 联合测序解析 |
| 结合单细胞核转录组测序(snRNA-Seq)技术,即可同步解析每个细胞核的基因表达信息与空间位置信息。 |
 |
|
|
| |
|
| ■ 微珠释放条形码吸附细胞核,无需形态学分割 |
| 传统空间转录组依赖复杂的图像分割算法界定细胞边界,而Trekker通过UV释放与物理结合机制,让条形码直接“绑定”细胞核,从而避免了形态学分割带来的误差,实现更精准的单细胞定位。 |
| |
| ■ 空间坐标单独解码,实现真正的单细胞空间组学分析 |
| 不同于单细胞数据+空间数据的后期整合的方法,Trekker的条形码与转录组信息在单细胞层面可以同步获得,消除了数据拼接的技术偏差,真正实现单细胞分辨率和空间定位的统一。 |
| |
| ■ 无缝兼容,支持10x Genomics、BD等单细胞平台 |
| 无需改造现有实验流程,Trekker可直接对接单细胞测序平台,在保留snRNA-Seq原始数据维度的基础上,额外赋予空间坐标信息,大大降低技术落地门槛。 |
|
|
| |
|
| ■ 成年小鼠脑细胞核的空间图谱构建 |
| 使用Trekker对来自25 μm成年小鼠大脑组织切片的27,275个细胞核进行空间定位。UMAP中的每个点代表一个单核,与空间图有1:1的对应关系,其中点通过来自snRNA-seq数据的基因表达模式进行颜色编码。 |
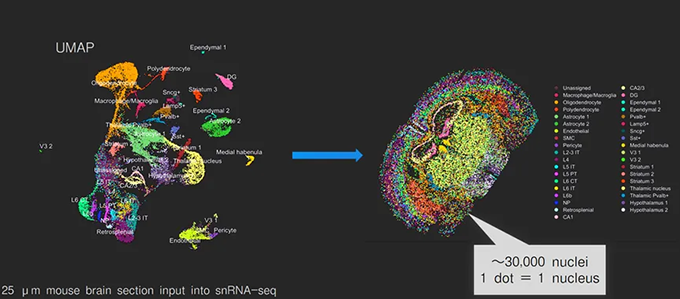 |
| 图A:单细胞UMAP转换为空间图谱 |
| |
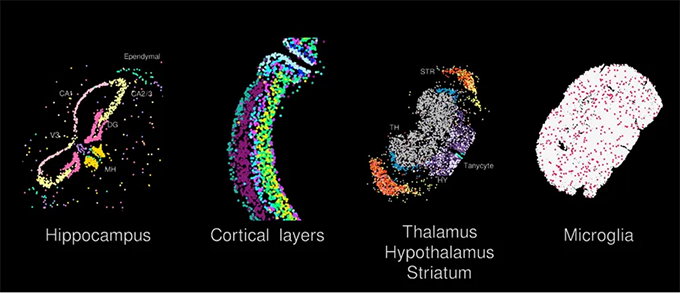 |
| 图B: 高基因覆盖率和单细胞数据揭示了细胞亚型形成的精细空间结构 |
| |
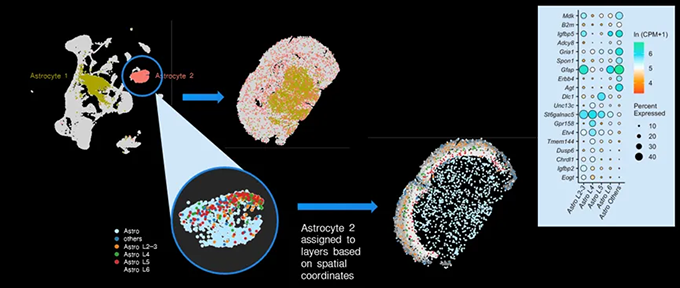 |
| 图C:揭示已知单细胞群体的空间异质性 |
| |
| ■ 生成高密度的细胞核空间图谱,同时保证单细胞数据的质量射 |
 |
| |
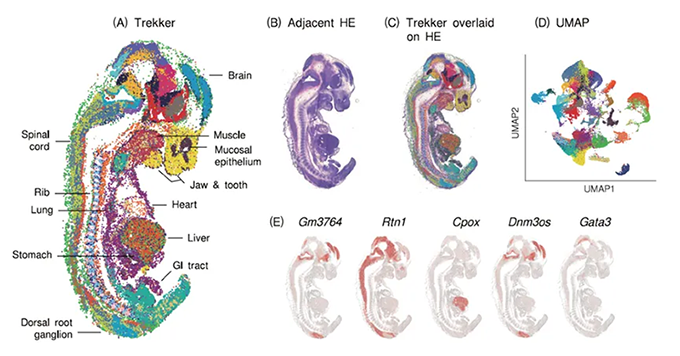
| 使用Trekker绘制第11天(E11)小鼠胚胎的细胞核的空间图谱。对25 μm的E11小鼠胚胎的组织切片上的57,545个细胞核使用Trekker Tile (10 mm×10 mm)进行空间分析。(A) 使用Trekker定位细胞核的空间图,每个点代表一个细胞核。(B) 相邻组织切片的H&E染色图像,显示出很高的一致性。(C) 通过ST align(连接图像和基因表达的工具)的H&E图像和Trekker空间图的叠加图像。(D) 单细胞分析的UMAP。每个点与图A中的点一一对应。(E) B中五种基因 (Gm3764、Rtn1、Cpox、Dnm3os、Gata3) 与H&E叠加的空间表达模式的图像。 |
|
|
| |
| 综上可见,Trekker的出现,打破“单细胞丢空间”“空间缺单细胞”的技术壁垒,在单细胞尺度直接捕获细胞的原生空间位置与基因表达信息。 |
 |
| |
| ☆ 产品规格 |
 |
| 更多详细Trekker信息,可通过4006518761,4006518769详询。 |
| |

