| 文献分享|单细胞TCR测序:解析肾细胞癌的免疫组库特征 |
| 前言: T细胞是获得性免疫反应的重要组成部分,其表面受体蛋白(T cell receptors, TCRs)发挥着识别抗原分子的作用。NGS是解析TCR多样性的主流技术,对揭示个体获得性免疫反应的奥秘提供了宝贵的技术支撑。而单细胞TCR-Seq技术一方面可以直接获得单细胞水平下克隆型的多样性信息;另一方面也可以直接对α-β链配对信息进行分析研究。本次分享的文献就涉及单细胞TCR-Seq的应用。 |
| 论文基本信息 ·论文题目:Characteristics, dynamic changes, and prognostic significance of TCR repertoire profiling in renal cell carcinoma patients ·发表杂志:The Journal of Pathology ·发表时间:2020-03-24 ·DOI:10.1002/path.5396 |
| 一、研究背景 |
| 肾细胞癌(Renal cell carcinoma,RCC)是起源于肾小管上皮的恶性肿瘤,占肾脏恶性肿瘤的 80%~90%。肾细胞癌的病因尚不明确,其发病与遗传、吸烟、肥胖等有关【1】。 虽然RCC被认为是一种高度免疫原性的癌症,但是免疫抑制时常发生。本文作者通过单细胞免疫组库,基于SMART技术和微流控技术,探究了RCC发展不同阶段和治疗前后肾癌的外周T细胞的TCRB的多样性的变化,并分析了TCRB多样性和RCC的动态关系。 |
| 二、分析思路 |
| ★ 研究方法 |
| ·为了研究TCRB的特征,首先从45例RCC患者和2例良性肾病患者获取87个PBMC样本,然后提取RNA并构建了基于SMART技术的TCRB测序文库。共获取到了10,072,661个TCRB克隆型,其中包括术前TCRB样本115,428±62,953(平均±SD)个和术后TCRB样本136,388±48,815(平均±SD)个。这里作者使用SMARTer Human scTCR a/b Profiling Kit构建单细胞TCRB文库。其中64个V基因和13个J基因均被检测到,并认为这种方法定量TCRB克隆型丰度是可行的。 ·为了解析TCRB多样性的意义,对28例患者术前和术后的56个PBMC样本进行了转录组测序。 ·为了验证结果,对一例晚期肾细胞癌患者的样本进行了单细胞RNA测序(scRNA,微流控方法)。 |
| ★ 研究发现 |
| ·TCRB的多样性和Native T细胞数量呈正相关(特别是CD4-和CD8-幼稚性T细胞)。接下来,选取162个与TCRB多样性正相关的基因进行GO分析,其中前20个terms主要是与生物合成与代谢过程有关 ;相反,GO分析结果显示TCRB多样性负相关的基因与炎症反应和凝血过程有关,特别是嗜中性粒细胞和血小板的激活。 ·结果显示,不同的肿瘤负荷对患者免疫状态产生不同的影响,四种类型的T细胞多样性指标一致表明良性和I期RCC患者的术前TCRB多样性要显著高于IV期患者; ·通过手术缓解了大多数肿瘤负荷后,IV期患者的TCRB多样性显著增加。结果表明,循环TCRB库能反映患者的免疫状态并预测预后。而且通过细胞减少肾切除术(cytoreductive nephrectomy, CN)在一定程度上减轻了晚期患者的免疫系统负担,可能为免疫治疗提供良好的机会。 |
| 使用的Takara产品:SMARTer Human scTCR a/b Profiling Kit |
| ■ 灵敏度高—整合SMART技术与5’RACE技术,可捕获TRA和TRB基因完整的V(D)J可变区,用于后续NGS分析 |
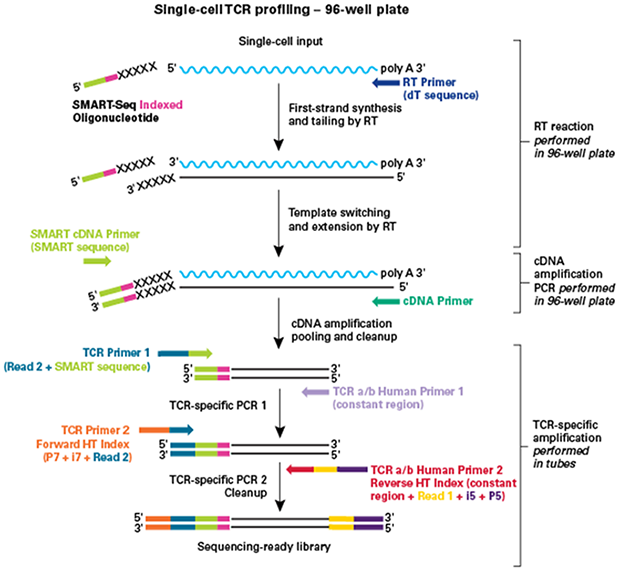 |
| ■ 易于使用—优化的index允许将96个细胞混合制成12个文库,并可以进一步混合在一个flow-cell lane中测序 |
| 实验孔板布局 |
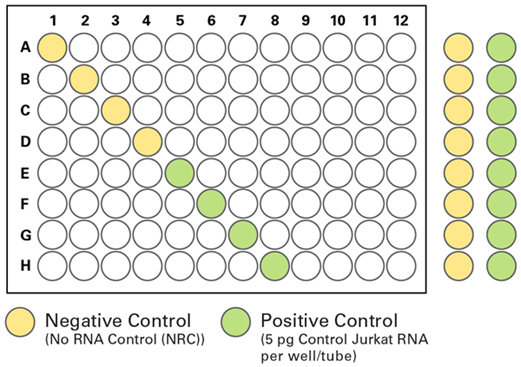 |
| 孔板外的阴性/阳性对照组用于测序文库的质量评估,孔板内的阴性/阳性对照孔用于克隆型鉴定分析时的阈值设定以及对潜在交叉污染的评估。严格的质控只为更好的测序结果。 |
| 添加SMART-Seq Indexed Oligo减少PCR偏差 |
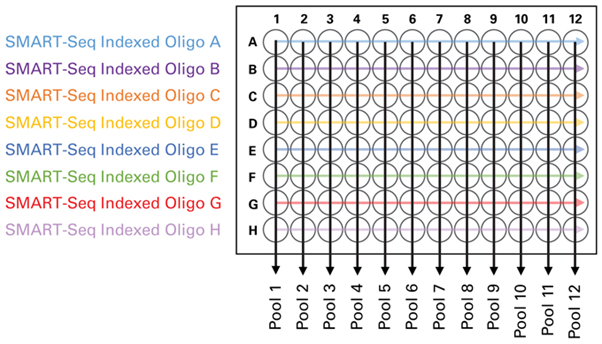 |
| 单细胞测序中涉及的文库数量更多,将每个单细胞文库做好标记对下游数据分析工作而言至关重要。通过SMART-Seq Indexed Oligo,可以在转录本cDNA第一链合成时,为同一细胞来源的转录本添加相同“身份标签”—— cell barcode。这样,孔板中同一列的8个细胞样品都有了自己的“身份标签”,将它们制备成12组混合文库进行V(D)J区序列的扩增,并添加上只属于自己组内的index标签序列。 |
| ■ 特异性好—完整reads,大部分reads可以比对到目标区域,提供准确链配对信息 |
| 实验案例 |
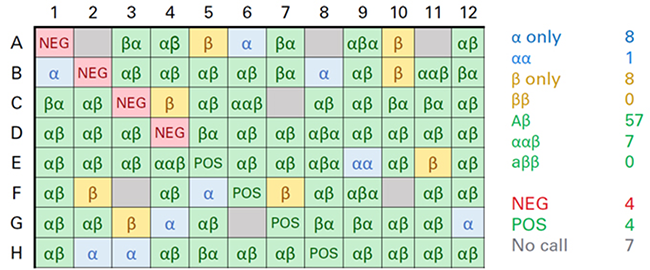
| 分选到96孔板中的CD4+ T细胞参照实验手册标准流程进行文库构建。使用Miseq测序仪进行测序,双端2×300 bp reads,600 cycles。测序数据分析中,8种SMART-Seq Indexed Oligo的拆分工作通过配套的 Human scTCR Demultiplexer软件可轻松完成。软件下载即用,无需安装。进一步的序列比对、克隆型分析通过开源软件MiXCR完成。 |
 |
| (数据来源于Takara Bio USA, Inc.) |
| 至少一条TCR链得到鉴定的比例是92%(81/88孔),α/β克隆型的鉴定比例是65%(57/88孔),同时鉴定到α和β链的克隆型比例是73%(64/88孔)。 |
| 写在最后,直接用于测序的文库构建流程、高效的混合测序策略、易用的数据分析软件,SMARTer Human scTCR a/b Profiling Kit帮助您快速上手单细胞TCR-Seq,获得高质量数据! |
| 参考信息: |
| 【1】 肾癌诊疗指南(2022年版) |
页面更新:2022-08-25 08:51:28