利用CRISPR筛选发现新冠病毒宿主因素 |
|||||||||||||
| 发现宿主因素对于了解新冠病毒发病机理以及识别新的治疗靶标是至关重要的。在病毒学研究领域,CRISPR筛选非常适用于病毒-宿主相互作用分析;确定对病毒入侵,复制和传播至关重要的宿主因素;确定新药物靶点并对其进行优先次序排序。利用sgRNA文库进行基因组规模的CRISPR/Cas9基因敲除筛选,可以帮助我们识别引起疾病反应的重要宿主因素。 |
|||||||||||||
| ■ 一步操作方案,轻松制备高质量sgRNA文库。 | |||||||||||||
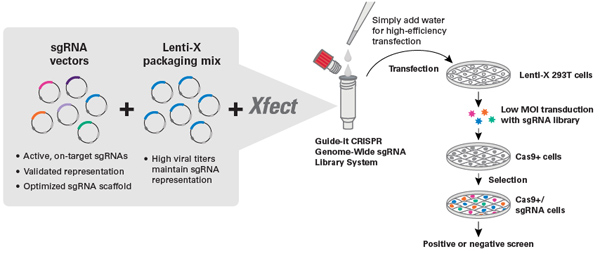
| Takara可以提供混合的慢病毒sgRNA文库系统Guide-it CRISPR Genome-Wide sgRNA Library System,用于在人细胞中进行表型敲除筛选。该文库系统基于Brunello文库(Doench et al. 2016; Doench et al. 2018)设计,包含超过76,000条向导RNA(sgRNA),靶向约19,000个基因,每个基因有四条高效的向导RNA以及非靶向sgRNA对照。这个扩增文库经过了充分验证,以可即时转染的冻干形式提供,可以让您轻松制备出高滴度慢病毒并建立全基因组CRISPR筛选。 | |||||||||||||
| 正如下图所示,我们的sgRNA文库系统,以单管冻干混合物形式提供,其中包含sgRNA载体,Lenti-X包装质粒和Xfect转染试剂。该系统通过高效的一步操作方案,可轻松完成转染和慢病毒制备过程。 |
|||||||||||||
 |
|||||||||||||
| CRISPR筛选后,可进行NGS分析,鉴定感兴趣的候选基因。Takara提供的NGS分析试剂盒Guide-it CRISPR Genome-Wide sgRNA Library NGS Analysis Kit包含以全基因组sgRNA文库转导的细胞集群为起始样品,制备Illumina测序用NGS文库所需要的引物和试剂。 |
|||||||||||||
| ■ 严紧的质量控制,以确保sgRNA代表性。 |
|||||||||||||
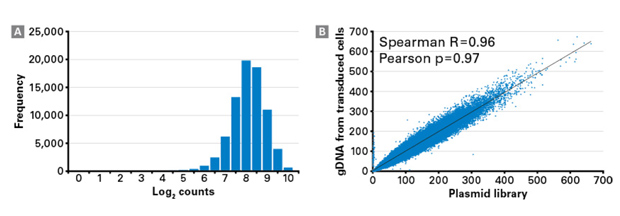
| 为了确保正确的sgRNA代表性,Takara对sgRNA文库进行了精心制作,测定了每一步操作的sgRNA代表性:包括文库合成、扩增、病毒制备和靶细胞转导步骤。下图Panel A展示了质粒文库中向导RNA的代表性。将基于Brunello的sgRNA库克隆到pLVXS-sgRNA-mCherry-hyg载体中并进行扩增。使用NGS技术分析验证质粒DNA文库sgRNA的代表性。检测结果显示,文库中超过90%的sgRNAs在10倍分布范围以内。图表中的柱形表示具有特定读段数的sgRNA数量。下图Panel B为质粒文库和转导细胞中sgRNA代表性的比较。以潮霉素为筛选标记,筛选出Cas9+/sgRNA+ A375细胞,提取基因组DNA并进行PCR扩增。测序分析PCR产物,检测PCR产物中每一个整合的sgRNA相对于起始质粒DNA文库的读段数。Spearman和Pearson相关性分析结果显示,这两者都具有很强的相关性,说明这个系统无论是在经过转导的细胞中还是筛选后的细胞中都可以有效保持sgRNA的代表性。 |
|||||||||||||
 |
|||||||||||||
|
|||||||||||||
| 使用全基因组CRISPR筛选进行病毒学研究发表的部分文献 |
|||||||||||||
| 1. Flint, M. A genome-wide CRISPR screen identifies N-acetylglucosamine-1-phosphate transferase as a potential antiviral target for Ebola virus. Nat. Comm. 285, (2019). 2. Gebre, M. et al. CRISPR-Cas9 Genetic Analysis of Virus-Host Interactions. Viruses 10, 55 (2018). 3. Han, J. Genome-wide CRISPR/Cas9 Screen Identifies Host Factors Essential for Influenza Virus Replication. Cell Rep. 23, 596-607 (2018). 4. Li, B. et al. Genome-wide CRISPR screen identifies host dependency factors for influenza A virus infection. Nat. Comm. 11, 164 (2020). 5. Marceau, C. D. Genetic dissection of Flaviviridae host factors through genome-scale CRISPR screens. Nature 535, 159-163 (2016). 6. Park, R. J. A genome-wide CRISPR screen identifies a restricted set of HIV host dependency factors. Nat. Genet. 49, 193-203 (2017). 8. Puschnik, A. S. et al. A CRISPR toolbox to study virus-host interactions. Nat. Rev. Microbiol. 15, 351-364 (2017). |
|||||||||||||
| 参考文献: |
|||||||||||||
| 1. Doench, J. G. et al. Optimized sgRNA design to maximize activity and minimize off-target effects of CRISPR-Cas9. Nat. Biotechnol. 34, 184-191 (2016). 2. Doench, J. G. et al. Am I ready for CRISPR A user's guide to genetic screens. Nat. Rev. Genet. 19, 67-80 (2018). |
|||||||||||||
页面更新:2020-05-15 10:13:29